| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | |
|---|---|---|---|---|---|---|---|---|---|---|
| I | Y | D | G | G | A | V | - | E | A | L |
| II | Y | D | G | G | I | L | V | E | A | L |
| III | F | E | G | G | I | L | V | E | A | L |
| IV | F | D | - | G | I | L | V | Q | A | V |
| V | Y | E | G | G | A | V | V | Q | A | L |
| Konsz. | y | d | G | G | A/I | V/L | V | e | A | l |

Összerendezés CLUSTAL-lal:
CLUSTAL W (1.60) multiple sequence alignment
hum-U1A ------MAVPETRPNHTIYINNLNEKIKKDELKKSLYAIFSQFGQILDILVSRSLKMRGQ
mse-U1A MATIATMPVPETRANHTIYINNLNEKIKKDELKKSLYAIFSQFGQILDILVSRIMKMRGQ
xla-U1A ------MSIQEVRPNNTIYINNLNEKIKKDELKKSLYAIFSQFGQILDILVSRNLKMRGQ
dme-U1A ---------MEMLPNQTIYINNLNEKIKKEELKKSLYAIFSQFGQILDIVALKTLKMRGQ
* * *************.*******************. . .*****
hum-U1A AFVIFKEVSSATNALRSMQGFPFYDKPMRIQYAKTDSDIIAKMKGTFVERDRKR-EKRKP
mse-U1A AFVIFKEVTSATNALRSMQGFPFYDKPMRIQYAKTDSDIIAKMKGTYVERDRKR-EKRKP
xla-U1A AFVIFKETSSATNALRSMQGFPFYDKPMRIQYSKTDSDIIAKMKGTFVERDRKRQEKRKV
dme-U1A AFVIFKEIGSASNALRTMQGFPFYDKPMQIAYSKSDSDIVAKIKGTFKERPKKVKPPKPA
******* **.****.***********.* *.*.****.**.***. ** .
hum-U1A KSQETPATKKAVQGGGATPVVGAVQGPVPGMPPMTQAPRIMHHMPGQPPYMPPPGMIPPP
mse-U1A KSQETPAAKKAVQGGAAAPVVGAVQ-PVPGMPPMPQAPRIMHHMPGQPPYMPPPGMIPPP
xla-U1A KVPEVQGVKNAMPGAALLPGVPGQMAAMQDMPGMTQAPRMMH-MAGQAPYMHHPGMMPPP
dme-U1A PGTDEKKDKKKK-----------------------------------------------P
. * *
hum-U1A GLAPGQIPPGAMPPQQLMPGQMPPAQPLSENPPNHILFLTNLPEETNELMLSMLFNQFPG
mse-U1A GLAPGQIPPGAMPPQQLMPGQMPPAQPLSENPPNHILFLTNLPEETNELMLSMLFNQFPG
xla-U1A GMAPGQMPPGGMPHGQLMPGQMAPMQPISENPPNHILFLTNLPEETNELMLSMLFNQFPG
dme-U1A SSAENSNP-----------------NAQTEQPPNQILFLTNLPEETNEMMLSMLFNQFPG
* . * . .*.***.*************.***********
hum-U1A FKEVRLVPGRHDIAFVEFDNEVQAGAARDALQGFKITQNNAMKISFAKK
mse-U1A FKEVRLVPGRHDIAFVEFDNEVQAGAARDALQGFKITQNNAMKISFAKK
xla-U1A FKEVRLVPGRHDIAFVEFDNEVQAGAARESLQGFKITQSNSMKISFAKK
dme-U1A FKEVRLVPNRHDIAFVEFTTELQSNAAKEALQGFKITPTHAMKITFAKK
********.********* .*.*..**...******* ..***.****
|
Összerendezés MultAlin-nal:
Multalin version 5.4.1
Copyright I.N.R.A. France 1989, 1991, 1994, 1996
Published research using this software should cite
Multiple sequence alignment with hierarchical clustering
F. CORPET, 1988, Nucl. Acids Res., 16 (22), 10881-10890
Symbol comparison table: blosum62
Gap weight: 12
Gap length weight: 2
Consensus levels: high=90% low=50%
Consensus symbols:
! is anyone of IV
$ is anyone of LM
% is anyone of FY
# is anyone of NDQEBZ
MSF: 289 Check: 0 ..
Name: hum-U1A Len: 289 Check: 9991 Weight: 0.67
Name: mse-U1A Len: 289 Check: 9174 Weight: 0.67
Name: xla-U1A Len: 289 Check: 7674 Weight: 0.67
Name: dme-U1A Len: 289 Check: 3145 Weight: 1.99
Name: Consensus Len: 289 Check: 255 Weight: 0.00
//
1 50
hum-U1A ......MAVP ETRPNHTIYI NNLNEKIKKD ELKKSLYAIF SQFGQILDIL
mse-U1A MATIATMPVP ETRANHTIYI NNLNEKIKKD ELKKSLYAIF SQFGQILDIL
xla-U1A ......MSIQ EVRPNNTIYI NNLNEKIKKD ELKKSLYAIF SQFGQILDIL
dme-U1A .........M EMLPNQTIYI NNLNEKIKKE ELKKSLYAIF SQFGQILDIV
Consensus ......m... E.rpN.TIYI NNLNEKIKK# ELKKSLYAIF SQFGQILDIl
51 100
hum-U1A VSRSLKMRGQ AFVIFKEVSS ATNALRSMQG FPFYDKPMRI QYAKTDSDII
mse-U1A VSRIMKMRGQ AFVIFKEVTS ATNALRSMQG FPFYDKPMRI QYAKTDSDII
xla-U1A VSRNLKMRGQ AFVIFKETSS ATNALRSMQG FPFYDKPMRI QYSKTDSDII
dme-U1A ALKTLKMRGQ AFVIFKEIGS ASNALRTMQG FPFYDKPMQI AYSKSDSDIV
Consensus vsr.$KMRGQ AFVIFKE..S AtNALRsMQG FPFYDKPMrI qYsKtDSDI!
101 150
hum-U1A AKMKGTFVER DRKR.EKRKP KSQETPATKK AVQGGGATPV VGAVQGPVPG
mse-U1A AKMKGTYVER DRKR.EKRKP KSQETPAAKK AVQGGAAAPV VGAVQ.PVPG
xla-U1A AKMKGTFVER DRKRQEKRKV KVPEVQGVKN AMPGAALLPG VPGQMAAMQD
dme-U1A AKIKGTFKER PKK....... .......... .......... ..........
Consensus AKmKGT%vER drKr.ekrk. k..e....k. a..g....p. v.........
151 200
hum-U1A MPPMTQAPRI MHHMPGQPPY MPPPGMIPPP GLAPGQIPPG AMPPQQLMPG
mse-U1A MPPMPQAPRI MHHMPGQPPY MPPPGMIPPP GLAPGQIPPG AMPPQQLMPG
xla-U1A MPGMTQAPRM MH.MAGQAPY MHHPGMMPPP GMAPGQMPPG GMPHGQLMPG
dme-U1A .......... .......... ......VKPP KPAPGTDEKK DKKKKPSSAE
Consensus mp.m.qapr. mh.m.gq.py m..pgm.pPP g.APGq.ppg .mp..qlmpg
201 250
hum-U1A QMPPAQPLSE NPPNHILFLT NLPEETNELM LSMLFNQFPG FKEVRLVPGR
mse-U1A QMPPAQPLSE NPPNHILFLT NLPEETNELM LSMLFNQFPG FKEVRLVPGR
xla-U1A QMAPMQPISE NPPNHILFLT NLPEETNELM LSMLFNQFPG FKEVRLVPGR
dme-U1A NSNP.NAQTE QPPNQILFLT NLPEETNEMM LSMLFNQFPG FKEVRLVPNR
Consensus #m.P.#p.sE #PPNhILFLT NLPEETNE$M LSMLFNQFPG FKEVRLVPgR
251 289
hum-U1A HDIAFVEFDN EVQAGAARDA LQGFKITQNN AMKISFAKK
mse-U1A HDIAFVEFDN EVQAGAARDA LQGFKITQNN AMKISFAKK
xla-U1A HDIAFVEFDN EVQAGAARES LQGFKITQSN SMKISFAKK
dme-U1A HDIAFVEFTT ELQSNAAKEA LQGFKITPTH AMKITFAKK
Consensus HDIAFVEFdn EvQagAAr#a LQGFKITq.n aMKIsFAKK
|
A dme-U1A szekvenciát (Drosophila fehérje) a Clustal és a MultAlin másképpen rendezi hozzá a többihez: Clustal két hézagot nyit, MultAlin csak egy hosszút. A MultAlinnál így több az azonosság, de a következtetés az, h. ez egy bizonytalan régió.
Melyik a legjobb módszer? Ezt nem lehet tudni, a helyes az, ha több módszert is alkalmazunk és az eredményekből konszenzust képezünk, manuálisan is igazítva
A1AA_HUMAN SLKYP....AI..MTER.KAA..AILALL.WVV.AL.VVSVGP.LLG...WKEPV..PPDE....RF A1AA_RAT SLKYP....AI..MTER.KAA..AILALL.WAV.AL.VVSVGP.LLG...WKEPV..PPDE....RF A1AB_CANFA SLQYP....TL..VTRR.KAI..LALLGV.WVL.ST.VISIGP.LLG...WKEPA..PNDD....KE A1AB_HUMAN SLQYP....TL..VTRR.KAI..LALLSV.WVL.ST.VISIGP.LLG...WKEPA..PNDD....KE A1AB_MESAU SLQYP....TL..VTRR.KAI..LALLSV.WVL.ST.VISIGP.LLG...WKEPA..PNDD....KE A1AB_RAT SLQYP....TL..VTRR.KAI..LALLSV.WVL.ST.VISIGP.LLG...WKEPA..PNDD....KE A1AC_BOVIN PLRYP....TI..VTQK.RGL..MALLCV.WAL.SL.VISIGP.LFG...WRQPA..PEDE.....T A1AC_HUMAN PLRYP....TI..VTQR.RGL..MALLCV.WAL.SL.VISIGP.LFG...WRQPA..PEDE.....T A1AC_RAT PLRYP....TI..VTQR.RGV..RALLCV.WVL.SL.VISIGP.LFG...WRQPA..PEDE.....T OAR_DROME PINYA....QK..RTVG.RVL..LLISGV.WLL.SL.LISSPP.LIG...W.NDW..PDEFT..SAT D1DR_CARAU PFRYE....RK..MTPR.VAF..VMISGA.WTL.SV.LISFIPVQLK...WHKAQ..PIGFL..EVN D1DR_FUGRU PFRYE....RK..MTPK.VAC..LMISVA.WTL.SV.LISFIPVQLN...WHKAQ..TASYVELNGT DADR_DIDMA PFRYE....RK..MTPK.AAF..ILISVA.WTL.SV.LISFIPVQLN...WHKARPLSSPDG..NVS ... A1AA_HUMAN ...............CGI..TE......................EAG...YA.....VF......SS A1AA_RAT ...............CGI..TE......................EVG...YA.....IF......SS A1AB_CANFA ...............CGV..TE......................EPF...YA.....LF......SS A1AB_HUMAN ...............CGV..TE......................EPF...YA.....LF......SS A1AB_MESAU ...............CGV..TE......................EPF...YA.....LF......SS A1AB_RAT ...............CGV..TE......................EPF...YA.....LF......SS A1AC_BOVIN ..............ICQI..NE......................EPG...YV.....LF......SA A1AC_HUMAN ..............ICQI..NE......................EPG...YV.....LF......SA A1AC_RAT ..............ICQI..NE......................EPG...YV.....LF......SA OAR_DROME ..............PCEL..TS......................QRG...YV.....IY......SS D1DR_CARAU ..............ASRR...DLPTDNC........DSSL....NRT...YA.....IS......SS D1DR_FUGRU ..............YAGD..LPPDNCD..........SSL....NRT...YA.....IS......SS DADR_DIDMA ..............SQDE...TMDNCD..........SSL....SRT...YA.....IS......SS ... A1AA_HUMAN V.CSFY...LPMAVIV.VMY.CRV.YVV....ARS..TTRSLEA.GVKR A1AA_RAT V.CSFY...LPMAVIV.VMY.CRV.YVV....ARS..TTRSL.....EA A1AB_CANFA L.GSFY...IPLAVIL.VMY.CRV.YIV....AKR..TTKNL.....EA A1AB_HUMAN L.GSFY...IPLAVIL.VMY.CRV.YIV....AKR..TTKNL.....EA A1AB_MESAU L.GSFY...IPLAVIL.VMY.CRV.YIV....AKR..TTKNL.....EA A1AB_RAT L.GSFY...IPLAVIL.VMY.CRV.YIV....AKR..TTKNL.....EA A1AC_BOVIN L.GSFY...VPLTIIL.VMY.CRV.YVV....AKR..ESRGLKS.GLKT A1AC_HUMAN L.GSFY...LPLAIIL.VMY.CRV.YVV....AKR..ESRGLKS.GLKT A1AC_RAT L.GSFY...VPLAIIL.VMY.CRV.YVV....AKR..ESRGLKS.GLKT OAR_DROME L.GSFF...IPLAIMT.IVY.IEI.FVA....TRR..RLRERA..RANK D1DR_CARAU L.ISFY...IPVAIMI.VTY.TQI.YRI....AQK..QIRRIS..ALER D1DR_FUGRU L.ISFY...IPVAIMI.VTY.TRI.YRI....AQK..QIRRIS..ALER DADR_DIDMA L.ISFY...IPVAIMI.VTY.TRI.YRI....AQK..QIRRIS..ALER |
597 darab G-fehérjéhez kapcsolt receptor szekvenciájának összerendezése (részlet!) a Pfam adatbázisból
OAR_DROME PINYAQ...KRTVGRVLLLISGVWLLSLLISSP.PLIG.WND.....WPDEFTSATP... D2DR_RAT PMLYNTR..YSSKRRVTVMIAIVWVLSFTISCP.LLFG.LNN...T.DQNE......... D3DR_RAT PVHYQHGTGQSSCRRVALMITAVWVLAFAVSCP.LLFG.FNT...TGDPSI......... DADR_RAT PFQYER...KMTPKAAFILISVAWTLSVLISFI.PVQLSWHKAK.PTWPLDGNFTSLEDT DBDR_RAT PFRYER...KMTQRVALVMVGLAWTLSILISFI.PVQLNWHRDKAGSQGQEGLLSNGTPW A1AB_RAT SLQYPT...LVTRRKAILALLSVWVLSTVISIG.PLLG.WKE......PAPNDDKE.... B1AR_RAT PFRYQS...LLTRARARALVCTVWAISALVSFL.PILMHWW.....RAESD.EARRCYND B2AR_HUMAN PFKYQS...LLTKNKARVIILMVWIVSGLTSFL.PIQMHWY.....RATHQ.EAINCYAN B2AR_RAT PFKYQS...LLTKNKARVVILMVWIVSGLTSFL.PIQMHWY.....RATHK.QAIDCYAK B3AR_RAT PLRYGT...LVTKRRARAAVVLVWIVSATVSFA.PIMSQWW.....RVGADAEAQECHSN 5HTA_RAT PIDYVN...KRTPRRAAALISLTWLIGFLISIP.PMLG.WRTPEDRSDPDA......... 5HTD_RAT ALEYSK...RRTAGHAAAMIAAVWAISICISIP.PLF..WRQ..ATAHEEMSD....... 5HT2_RAT PIHHSR...FNSRTKAFLKIIAVWTISVGISMPIPVFG.LQDDSKVFKEGS......... ... OAR_DROME ............CELTSQRG.................YVIYSSLGSPFIPLAIMTIVYIEIFVATRRRLRERARANK D2DR_RAT ............CIIANPA..................FVVYSSIVSFYVPFIVTLLVYIKIYIVLRKRRKR...... D3DR_RAT ............CSISNPD..................FVIYSSVVSFYVPFGVTVLVYARIYIVLRQRQRK...... DADR_RAT ED........DNCDTRLSRT.................YAISSSLISFYIPVAIMIVTYTSIYRIAQKQIRR...... DBDR_RAT EEGWELEGRTENCDSSLNRT.................YAISSSLISFYIPVAIMIVTYTRIYRIAQVQIRR...... A1AB_RAT ............CGVTEEPF.................CALFCSLGSFYIPLAVILVMYCRVYIVAKRTTKN...... B1AR_RAT PK..........CCDFVTNRA................YAIASSVVSFYVPLCIMAFVYLRVFREAQKQVKK...... B2AR_HUMAN ET..........CCDFFTNQA................YAIASSIVSFYVPLVIMVFVYSRVFQEAKRQLQK...... B2AR_RAT ET..........CCDFFTNQA................YAIASSIVSFYVPLVVMVFVYSRVFQVAKRQLQK...... B3AR_RAT PR..........CCSFASNMP................YALLSSSVSFYLPLLVMLFVYARVFVVAKRQRRF...... 5HTA_RAT ............CTISKDHG.................YTIYSTFGAFYIPLLLMLVLYGRIFRAARFRIRK...... 5HTD_RAT ............CLVNTSQIS................YTIYSTCGAFYIPSILLIILYGRIYVAARSRILN...... 5HT2_RAT ............CLLADDN..................FVLIGSFVAFFIPLTIMVITYFLTIKSLQKEATL...... ... |
687 G-fehérjéhez kapcsolt receptor szekvenciájának összerendezése (részlet!) a PRINTS adatbázisból

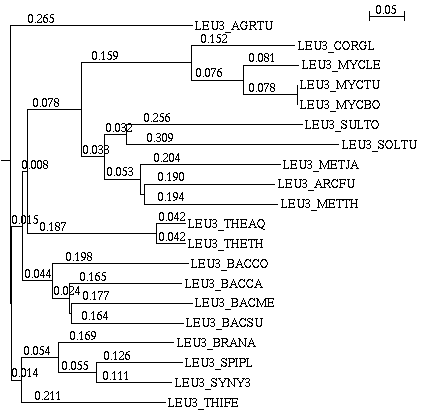
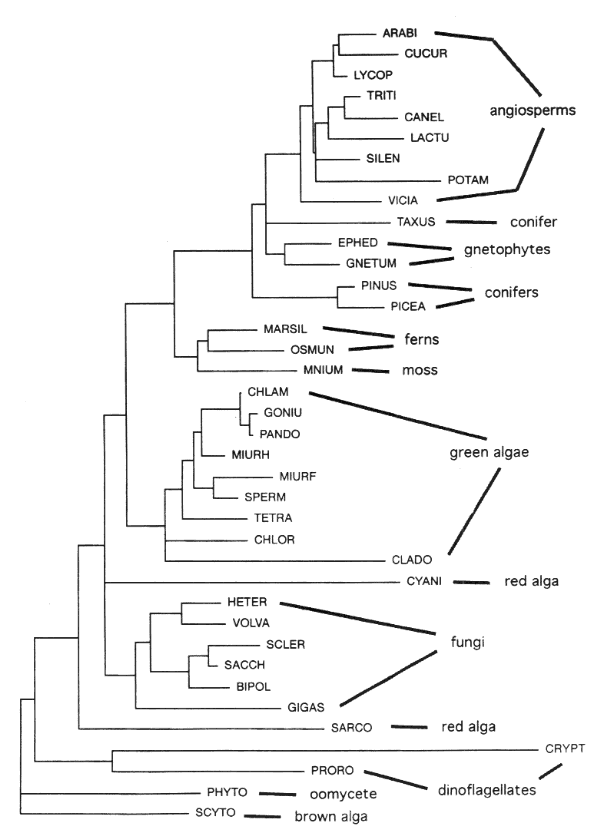
Gyökeres fa
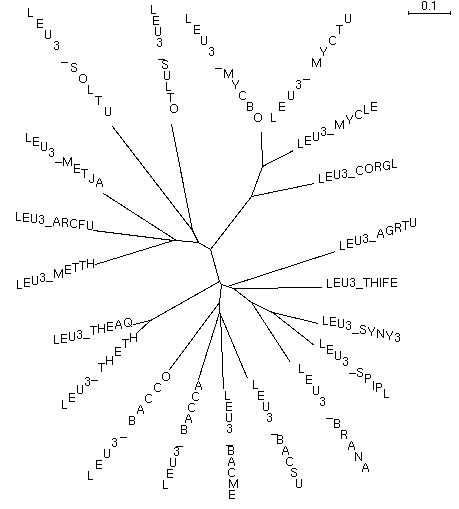
Gyökértelen fa
116 | 1 ARABI GCGCCC 2 LYCOP .....C 3 triti .....C 4 LACTU .....C 5 SILEN .....C 6 vicia .....C 7 CANEL .....C 8 potam .....C 9 ephed .....C 10 gnetu .-...C 11 PINUS .....C 12 PICEA .....C 13 TAXUS .GC..G 14 marsi .-...C 15 osmun .-...C 16 mnium .....C 17 CHLAM ....TC 18 SPERM ....TC 19 TETRA ....TC 20 CHLOR ....TC 21 CLADO ....TC | 122-144 ---CAAGCCTTCT-GGCCG---- ---GAAGCCATTT-GGCCG---- ---GAGGCCACTC-GGCCG---- ---GAAGCCATCC-GGCTG---- ---GAAGC--TTC-GGCTG---- ---GATGCCATTA-GGTTG---- ---GAGGCCACTA-GGCTG---- ---TAAGCTTCCG-GGCCG---- ---GAAGCC--TC-CGCCA---- TCCG-AGCC--TA-GGCCG---- ---GAGGCC--TC-GGTCG---- ---GAGNCC--TC-GGTCG---- ---GAG-C---TC-GGCCG---- ---GAGGC---TC-GTCCG---- ---GCGGC---TC-GTCCA---- ---GAGGC---TC-GTCCG---- ---GAGGC--TTC-GGCCA---- ---GAGGC--TTC-GGCCG---- ---GAGGC--CTC-GGCCA---- ---GAGAC--CTC-GGTCA---- ---AAGTC--TAC-GGACT---- | 155 | AGGGCACGTCT A.......... A.......C.. A.......C.. A.......... A.......... A....T..C.. A.....A.C.. A.......... A.......... A.......... A.......... A.......... A.......... A.....T.C.. A.....TT..C A.A...T.... A.A...T..T. A.A.....C.. A.A...T.... T.A...T.... |
22 HETER .....C 23 VOLVA ....TC 24 SCLER .....C 25 sacch .....C 26 BIPOL .....C 27 GLOMU ....TC | TTT--GGT-ATT----CCGA--- TTT--GGCCATT----CCGA--- CTT--GGT-ATT----CCGG--- CTT--GGT-ATT----CCAG--- TTT--GGT-ATT----CCAA--- CCT--GGT-ATT----CCGG--- | A..-....C.. A.A...T.C.. G.....T.C.. G.....T.C.. A.....T.C.. G.A.T.T.C.. |
28 CYANI ....TT 29 SARCO ....TC 30 PHYTO ..A.TT 31 SCYTO ...-TT 32 crypt .---CT 33 PRORO ....TT | TC--AGGAGAATTTTATTTTCCT GC---GGTAA-TC--------CT CCG--GGTTAGTC---CTG---- CCG--GGTTAGTC---CTG---- CC---AGC--TGA---CT----- TCG--GGATATCC---CTG---- | G.A........ GCA.--T.... G.A.T.T.C.. G.A...T.CT. ------T...A AA....T.C.. |
Kiinduló összerendezés, a szekvenciák az organizmusok rendszertani hovatartozása szerinti sorrendben vannak. A középső részben lévő szekvenciák három csoportra különülnek el (zöld növények, gombák, egysejtűek), s kevéssé hasonlítanak, összerendezésük önkényes. (A pontok itt az oszlop tetején jelölttel azonos bázist jelentenek.)
Ebből állítjuk elő "sebészkedés" révén a filogenetikai analízisre előkészített összerendezést:
116 | 1 ARABI GCGCCC 2 LYCOP GCGCCC 3 triti GCGCCC 4 LACTU GCGCCC 5 SILEN GCGCCC 6 vicia GCGCCC 7 CANEL GCGCCC 8 potam GCGCCC 9 ephed G-GCCC 10 gnetu G-GCCC 11 PINUS GCGCCC 12 PICEA GCGCCC 13 TAXUS GGCCCG 14 marsi G-GCCC 15 osmun G-GCCC 16 mnium GCGCCC 17 CHLAM GCGCTC 18 SPERM GCGCTC 19 TETRA GCGCTC 20 CHLOR GCGCTC 21 CLADO GCGCTC | 122-144 ???CAAGCCTTCT?GGCCG???? ???GAAGCCATTT?GGCCG???? ???GAGGCCACTC?GGCCG???? ???GAAGCCATCC?GGCTG???? ???GAAGC?-TTC?GGCTG???? ???GATGCCATTA?GGTTG???? ???GAGGCCACTA?GGCTG???? ???TAAGCTTCCG?GGCCG???? ???GAAGCC?-TC?CGCCA???? TCCG?AGCC?-TA?GGCCG???? ???GAGGCC?-TC?GGTCG???? ???GAG?CC?-TC?GGTCG???? ???GAG-C?-?TC?GGCCG???? ???GAGGC??-TC?GTCCG???? ???GCGGC??-TC?GTCCA???? ???GAGGC??-TC?GTCCG???? ???GAGGC?-TTC?GGCCA???? ???GAGGC?-TTC?GGCCG???? ???GAGGC?-CTC?GGCCA???? ???GAGAC?-CTC?GGTCA???? ???AAGTC?-TAC?GGACT???? | 155 | AGGGCACGTCT AGGGCACGTCT AGGGCACGCCT AGGGCACGCCT AGGGCACGTCT AGGGCACGTCT AGGGCTCGCCT AGGGCAAGCCT AGGGCACGTCT AGGGCACGTCT AGGGCACGTCT AGGGCACGTCT AGGGCACGTCT AGGGCACGTCT AGGGCATGCCT AGGGCATTTCC AGAGCATGTCT AGAGCATGTTT AGAGCACGCCT AGAGCATGTCT TGAGCATGTCT | [ 122'-141' ] ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? |
22 HETER GCGCCC 23 VOLVA GCGCTC 24 SCLER GCGCCC 25 sacch GCGCCC 26 BIPOL GCGCCC 27 GLOMU GCGCTC | ??????????????????????? ??????????????????????? ??????????????????????? ??????????????????????? ??????????????????????? ??????????????????????? | AGG-CACGCCT AGAGCATGCCT GGGGCATGCCT GGGGCATGCCT AGGGCATGCCT GGAGTATGCCT | TTT??GGT-ATT????CCGA TTT??GGCCATT????CCGA CTT??GGT-ATT????CCGG CTT??GGT-ATT????CCAG TTT??GGT-ATT????CCAA CCT??GGT-ATT????CCGG |
28 CYANI GCGCTT 29 SARCO GCGCTC 30 PHYTO GCACCT 31 SCYTO GCG-TT 32 crypt G??-CT 33 PRORO GCGCTT | ??????????????????????? ??????????????????????? ??????????????????????? ??????????????????????? ??????????????????????? ??????????????????????? | GGAGCACGTCT GCAG-?TGTCT GGAGTATGCCT GGAGCATGCTT ??????TGTCA AAGGCATGCCT | ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? ???????????????????? |
A filogenetikai elemzéshez előkészített összerendezés.
Ez az összerendezés a következő módosítások által keletkezett a kiinduló összerendezésből:
Ezek meghatározásának két módja:
A C G T
A - 2 1 2
C 2 - 2 1
G 1 2 - 2
T 2 1 2 -
A pontszámok a csere költségét mutatják, purinbázis pirimidinbázisra való cseréje (transzverzió) ritkább, ezért költségesebb, mint a purin-purin és a pirimidin-pirimidin csere (tranzíció).
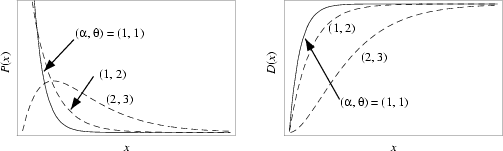
Gamma eloszlás. Bal oldalon a valószínűségeloszlás sűrűségfüggvénye, jobb oldalon az eloszlásfüggvény, mindkettő a két paraméter (alfa, théta) több lehetséges értéke mellett.
Melyik helyettesítési modellt válasszuk? A kevés paraméterrel dolgozó modellek jobban alkalmazhatóbbak, megbízhatóbbak, a túlságosan leegyszerűsített modellek viszont hibás eredményt adhatnak. Fontos a tranzíciós és a transzverzió megkülönböztetése és a mutációgyakoriság heterogenitásának figyelembe vétele. Gondosan kell kiválasztani az adott adatokhoz legjobban illeszkedő evolúciós modellt.
A faépítő módszerek kétféle osztályozása:
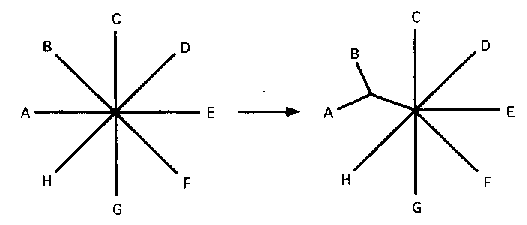
Kétféle módszer: