2. Adatbázisok
Vázlat:
- Adatbázisok általában
- Nukleinsavszekvencia-adatbázisok
- Elsődleges DNS-szekvencia-adatbázisok
- Specializált adatbázisok
- Fehérjeszekvencia-adatbázisok
- Elsődleges fehérjeszekvencia-adatbázisok
- Összetett fehérjeszekvencia-adatbázisok
- Másodlagos és harmadlagos (szekvenciamintázat-)adatbázisok
- Térszerkezeti adatbázisok
- Fehérjecsaládok adatbázisai
- Klaszterezés
- Szekvenciacsaládok adatbázisai
- Szerkezeti családok adatbázisai
Adatbázisok általában
Rengeteg adatbázis, a következő részterületeken:
- DNS-szekvenciák
- Összehasonlító genomadatbázisok
- Génexpressziós adatok
- Génazonosítás és génszerkezet
- Genetikai és fizikai genomtérképek
- Genomok
- Intermolekuláris kölcsönhatások
- Anyagcsere- és szabályozási útvonalak
- Mutációk
- Betegségek
- Fehérjék
- Fehérjeszekvencia-mintázatok
- Proteomelemzés
- RNS-szekvenciák
- Térszerkezetek
- Gyógyszerek
- ...stb.
Már adatbázisok adatbázisa (katalógusa) is van: DBCAT
A Nucleic Acids Research folyóirat minden évben Adatbázis-különszámot ad
ki. Ebben van egy hasznos adatbázislista.
Nukleinsavszekvencia-adatbázisok
Elsődleges DNS-szekvencia-adatbázisok
- A következők:
- GenBank (USA, National Center for Biotechnology Information)
- EMBL (Európa, European Bioinformatics Institute)
- DDBJ (DNA Data Base of Japan, Japán, National Institute of
Genetics)
- Együtt: International Nucleotide Sequence Database Collaboration
- Mindhárom adatbázis fenntartója gyűjti a szekvenciaadatokat:
- közvetlenül a szekvenálást végző kutatóktól
- az irodalomból
- szabadalmakból
- a nagy genomszekvenálási projektekből
- Együttműködnek, új adataikat naponta kicserélik egymás között (ezért
ugyanaz van bennük)
- Méretei:
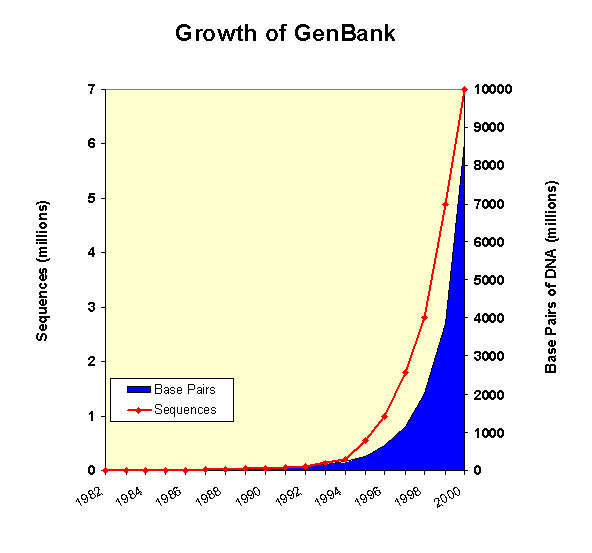
2001 augusztus: kb. 12,8 millió szekvencia, ezekben 13,5 milliárd bázis. 14
havonként megduplázódik.
- A GenBank 17 kisebb szekcióból (division) áll, melyeknek hárombetűs rövidítése
van:
| Szekció | Milyen szekvenciákat tartalmaz | Megjegyzés
|
|---|
| PRI | Főemlősök (Primates) | Az élővilág egyes
csoportjai szerinti szekciók
|
| ROD | Rágcsálók (Rodents)
|
| MAM | Egyéb emlősök (Mammals)
|
| VRT | Egyéb gerincesek (Vertebrates)
|
| INV | Gerinctelenek (Invertebrates)
|
| PLN | Növények (Plant), gombák, moszatok
|
| BCT | Baktériumok (Bacteria)
|
| VRL | Vírusok (Viral)
|
| PHG | Bakteriofágok (phage)
|
| SYN | Szintetikus szekvenciák (synthetic) | Egyéb
kategóriák
|
| UNA | Annotáció nélküliek (unannotated)
|
| EST | Expressed Sequence Tag-ek
|
| PAT | Szabadalmaztatott (patent)
|
| STS | Sequence Tagged Sites
|
| GSS | Genome Survey Sequences
|
| HTG | High Throughput Genomic Sequences
|
| HTC | High Throughput cDNA Sequences
|
- STS: Olyan, rövid szekvenciarészletek (<400 bp), amelyek a
genomban csak egy helyen, egy bizonyos pozícióban találhatóak meg.
Markerként, tájékozódási pontként használatosak.
- EST: Olyan STS-szekvenciák, amelyeket cDNS részleges szekvenálásával nyertek
(komplementer DNS: az mRNS DNS-re átírt változata). Felhasználhatóak egy
gén azonosításához, térképezéséhez, klónozásához.
- GSS: mint az EST, de a genomból nyert szekvenciák (nem feltétlenül
egyedülállóak)
- HTG: a nagy genomszekvenálási projektekből nyert, hosszabb, még nem
véglegesített szekvenciák. Négy kategória:
- Phase 0: klónokból véletlenszerűen nyert szekvenciarészletek
- Phase 1: nem véglegesített szekvencia, rendezetlen, irányítatlan
darabokból állhat, hézagokkal
- Phase 2: már rendezett, irányított darabok, hézagokkal
- Phase 3: véglegesítendő szekvencia, átkerül a megfelelő
rendszertani szekcióba
- HTC: nagy szekvenálási projektekből nyert cDNS szekvenciák
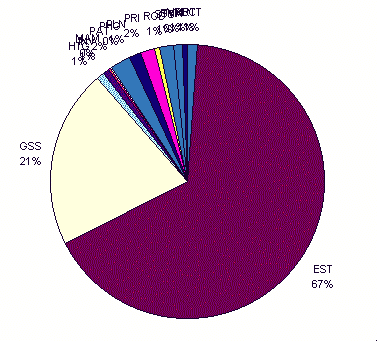
- A szekciókban lévő szekvenciák számának megoszlása (2000 október):
- A szekciókban lévő bázisok számának megoszlása:
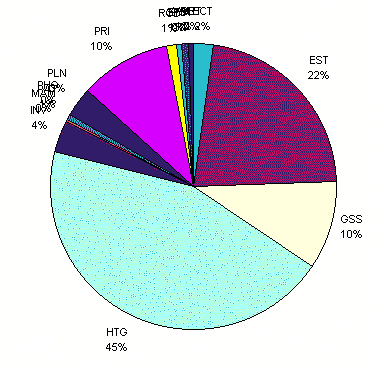
A szekvenciák száma szerinti eloszlásban az EST-k vannak többségben
(67%), de a bázisok száma szerint nem, mert rövidek.
A GenBank fájlok felépítése ("flat file")
Példa
Kulcsszavak és alkulcsszavak:
| Kulcsszó | Magyarázat, alkulcsszavak
|
|---|
| LOCUS | Rövid címke (pl. itt HUMCYCLOX: humán ciklooxigenáz),
bázisok száma (itt: 3387), forrás (itt: mRNS), szekció (itt:
PRI=főemlősök), beküldés dátuma
|
| DEFINITION | A szekvencia rövid leírása (pl. minek a génje)
|
| ACCESSION | Azonosítószám: a szekvencia egyedi azonosítója
|
| VERSION | Azonosítószám.verziószám: javítás esetén az első nem
változik, csak a második. GI (geninfo identifier): az aktuális verzióhoz
rendelt további azonosítószám
|
| KEYWORDS | kulcsszavak, a szerző adhatja meg őket, bár a
GenBanknál nem szeretik (nincs szabványosítva)
|
| SOURCE | Milyen szövetből származik a
DNS
| ORGANISM | Forrásorganizmus rendszertani
besorolása |
|
| REFERENCE | irodalmi hivatkozás az egyes
szekvenciaszakaszokra
| AUTHORS | szerzők |
| TITLE | cím | | JOURNAL | folyóirat |
| MEDLINE | Medline azonosító |
|
| FEATURE | "Feature table": a szekvencia tulajdonságainak
részletes leírása, szakaszonként, a szakaszhatárok megadásával
| source | forrásorganizmus (faj, szövet, sejttípus, hivatkozás a
taxonómiai adatbázisra (db_xref)
| | 5'UTR | nem transzlálódó szakasz az 5' végen
| | gene | a tulajdonképpeni gén
| | CDS | a kódoló szekvencia (coding sequence), megfelelő
kereszthivatkozások a fehérjeadatbázisra, fordítás
| | sig_peptide | szignálpeptid
| | mat_peptide | érett peptid (a szignálpeptid lehasadása után
maradó rész)
| | 3'UTR | nem transzlálódó szakasz a 3' végen
| | polyA_signal | poliadenilációs szakasz
| |
| BASE COUNT | Melyik bázisból hány van
|
| ORIGIN | A szekvencia helye a genomon belül (ha ismert)
|
| | nukleotidszekvencia
|
| // | rekord vége
|
Specializált nukleinsavszekvencia-adatbázisok
- dbEST, dbSTS, dbGSS (az NCBI-nél): a GenBankban is meglévő szekvenciák
adatbázisai, de bővebb információt is szolgáltatnak az egyes szekvenciákról
- Genomadatbázisok: Ensembl (emberi genom), SGD (élesztő genomja), ACeDB (A Canenorhabditis
elegans DataBase, C. elegans genomja), stb. Fizikai géntérképekkel, stb. (Részletesen majd a genomikai
előadásban)
- COGs: Clusters of Orthologous Groups of proteins: fehérjéket kódoló
DNS-szekvenciák filogenetikai rendszerezése. 44 organizmus teljes genomja
alapján készült. Az ortológokat egy csoportba sorolja. (Részletesebben majd a genomikai
előadásban.)
Fehérjeszekvencia-adatbázisok
Elsődleges fehérjeszekvencia-adatbázisok
- 1960-as években alapozta meg Margaret Dayhoff a National Biomedical
Research Foundationnál (USA)
- 1988 óta: PIR-International konzorcium tartja fenn (NBRF, JIPID [Japanese
International Protein Information Database], MIPS [Munich Information Center for
Protein Sequences])
- Mintegy 250 000 szekvencia, ezekben 80 millió aminosav (2001 szeptember)
- 4 szekció:
- PIR1: teljesen klasszifikált, annotált szekvenciák
- PIR2: előzetes szekvencia, részlegesen ellenőrzött és klasszifikált
(ide tartozik az adatbázis legnagyobb része)
- PIR3: egyáltalán nem ellenőrzött, nem osztályzott, nem annotált (pár
száz db)
- PIR4: a természetben nem előforduló, nem expresszálódó szekvenciák (pl.
pszeudogének transzlációi, hibás transzlációk, szintetikus szekvenciák) (pár
száz db)
- A szekvenciákat szekvenciaazonosság alapján családokba sorolja.
- 1986-ban született a genfi egyetem és az EMBL együttműködéseként,
1994-től a hinxtoni EBI (European Bioinformatics Institute) vette át, majd
1998-tól a SIB (Swiss Institute of Bioinformatics) is beszállt
- Mintegy 101 000 szekvencia (37 millió aminosav) kb. 7200 fajból
(2001 szeptember)
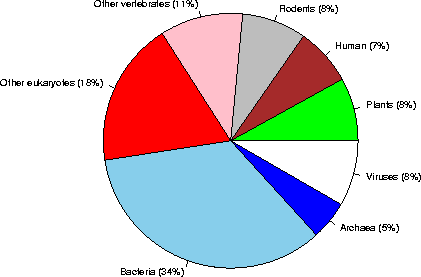
A szekvenciák megoszlása az élőlények csoportjai szerint
- Fő törekvések:
- nagyon magas szintű és részletes annotáció minden
fehérjéhez (funkció, doménszerkezet, másodlagos és negyedleges szerkezet, poszttranszlációs módosítások,
változatok, a fehérjével összefüggő betegségek, stb.)
- minimális redundancia
- bőséges kereszthivatkozások más adatbázisokra
A SWISS-PROT rekordok felépítése
Példa
| Kód | Jelentés
|
|---|
| ID | Identifier: azonosító. Ált. FEHÉRJE_FORRÁSORGANIZMUS alakú. Ez
változhat!
|
| AC | Accession number: a szekvenciához rendelt azonosító kód,
sohasem változik
|
| DT | Date: létrehozás és módosítások dátumai
|
| DE | Description: leírás (minek a szekvenciája)
|
| GN | Gene name: a gén neve
|
| OS | Organism species: milyen fajból származik
|
| OC | Organism classification: a faj rendszertani besorolása
|
| RN | Reference number: hivatkozás sorszáma
|
| RP | Reference position: a hivatkozással kapcsolatos
szekvenciaszakasz
|
| RA | Reference authors: szerzők
|
| RL | Reference location: folyóirat, kötet, oldal
|
| RT | Reference title: cím
|
| RX | Reference cross-references: kereszthivatkozások pl. Medline
|
| CC | Comments: megjegyzések. Információk a funkcióról,
poszttranszlációs módosításokról, szövetspecificitásról, sejtbeli
elhelyezkedésről, fehérjecsaládhoz tartozásról
|
| DR | Database cross-references: kereszthivatkozások más adatbázisokra
|
| KW | Keywords: kulcsszavak
|
| FT | Feature table: információk az egyes szekvenciaszakaszokról
(domének, transzmembrán szakaszok, másodlagos szerkezet, diszulfidhidak,
kötőhelyek, módosított oldalláncok, szignálszekvenciák, stb., a megbízhatóság jelölésével)
|
| SQ | Szekvencia hossza, számított molekulatömeg, ellenőrző összeg
|
| | maga a szekvencia
|
| // | rekord vége
|
TrEMBL (translated EMBL)
- A SWISS-PROT társadatbázisa, az EMBL-ben lévő összes CDS (kódoló
szekvencia) lefordítása aminosavszekvenciára, számítógépesen annotálva
- Egy részük fokozatosan átkerül a SWISS-PROTba
- Mintegy 480 000 szekvencia (2001 szept.)
- A Protein Data Bank térszerkezeti adatbázisból kivont szekvenciákat
tartalmazza
- Csak a PDB-ben ténylegesen szerkezettel rendelkező aminosavak
- Bőséges annotáció (másodlagos szerk., felbontás, stb.)
Melyik szekvenciaadatbázist használjuk?
- NRL_3D a legkisebb, de van hozzá szerkezeti információ
- PIR(1-4) a legnagyobb, de az annotáció sokszor szegényes
- SWISS-PROT kiválóan annotált, de kevés szekvencia van benne
Két megoldás:
- Mindegyiket használjuk
- Összetett adatbázist használunk
Összetett fehérjeszekvencia-adatbázisok
Céljuk: egyesíteni az elsődleges fehérjeszekvencia-adatbázisokban lévő
szekvenciákat, a redundanciák kiszűrésével. Több ilyen is van:
- Forrás: PDB, SWISSPROT, PIR, GenPept (a GenBank transzlációja), SPupdate
(a SWISSPROT hetenkénti frissítései), GenPeptupdate (a GenPept naponkénti
frissítései)
- Átfogó és naprakész
- De: Redundáns! Csak a teljes azonosságok vannak kiszűrve.
Polimorfizmusok és kisebb szekvenálási hibák miatt
ugyanaz a fehérje többször is szerepelhet; egyes rekordok más rekordokban
lévő fragmentumok egyesítései, stb.
- NCBI fehérjeadatbázisa
- Forrás: SWISSPROT (legnagyobb prioritással), PIR, GenPept, NRL_3D
- Nem naprakész, csak 6-8 hetenként frissítik
- Kb. 280 000 szekvencia
- Kevésbé redundáns az NRDB-nél (egy aminosavban eltérő szekvenciákat is
azonosnak vesz a szűréskor), de mégis van valamennyi redundancia
SWISS-PROT + TrEMBL
- ExPASy-nál együttesen kereshető a két adatbázis
- Naprakész
- Kevésbé redundáns, mint az OWL és az NRDB, de még így is kb. 30%-ban
redundáns
Melyik összetett adatbázist használjuk?
- NRDB naprakész, de elég redundáns
- OWL kevésbé redundáns, de nem naprakész
- SWISSPROT+TrEMBL nem elég átfogó
- Legbiztosabb, ha mindegyiket használjuk, bár ez ellentétes a céljukkal!
Másodlagos és harmadlagos (szekvenciamintázat-)adatbázisok
Szekvenciamintázat-adatbázisok
- Az elsődleges adatbázisokban (a harmadlagosoknál a másodlagosokban lévő
információk) lévő szekvenciák alapján többszörös szekvenciaösszerendezéseket
készítünk. Ekkor láthatóvá válnak a konzerválódott régiók, a
motívumok. Ezekből vezetik le a másodlagos, ill. a harmadlagos
adatbázisokat.
- Számos adatbázis, részletesebben majd külön előadásban.
- Szekvenciaanalízisben nagy jelentőségük van
Térszerkezeti adatbázisok
- Brookhaven National Lab-ból átkerült a Research Collaboratory for
Structural Bioinformatics-hoz
- Kb. 16 000 szerkezet, ebből kb. 15 000 fehérje, a többi
nukleinsav és szénhidrát
- Erősen redundáns (ugyanaz a fehérje sokszor szerepelhet: különböző
szubsztrátokkal és kristályosítási körülményekkel, mutánsok, stb.)
- Röntgen- és NMR-szerkezetek
A konnektivitás problémája
- A szekvencia megadja a molekula konnektivitását is (melyik atom
melyikhez van kötve)
- Szerkezet-feldolgozó programok két módon reprodukálják a konnektivitást:
- Kémiai szabályok alapján: vegyértékek, kötéshosszak alapján.
Hátrány: pontatlan koordináták esetén hibás konnektivitás adódik
- Molekulacsoport-könyvtárakkal: külön tároljuk az összes
aminosav, kismolekula, stb. rendes szerkezetét a konnektivitásokkal együtt.
Nem adódhat hibás konnektivitás.
Fájlformátumok
- Klasszikus PDB formátum: Példa
| Kulcsszó | Magyarázat
|
|---|
| HEADER | Fejléc, a molekula besorolása
|
| COMPND | A molekula megnevezése
|
| SOURCE | Forrásorganizmus
|
| AUTHOR | Szerzők
|
| REVDAT | Frissítési dátumok
|
| JRNL | Az irodalmi hivatkozás
|
| REMARK | Megjegyzések: további irodalmi hivatkozások, felbontás, a
finomítás módszerei, megjegyzések a szerkezethez, javítások
|
| SEQRES | A szekvencia. Nem mindig ugyanaz, mint a koordinátáknál
megadott szekvencia! Itt a valódi biológiai szekvencia van.
|
| HELIX, SHEET | Másodlagos szerk.
|
| SSBOND | Diszulfidhidak
|
| SITE | Megjelölt helyek, pl. kötőhelyek, aktív helyek
|
| CRYST1 | Elemi cella adatai
|
| MTRIX | Másik alegység generáláshoz szükséges mátrix
|
| ATOM, HETATM | Az egyes atomok adatai. Sorszám, atom neve, alternatív
pozíció jelzője, oldallánc neve,
lánc betűjele, oldallánc sorszáma, beszúrási kód, koordináták, occupancy,
hőmérsékleti faktor, lábjegyzet száma
|
| TER | Láncvég
|
| CONECT | Összekötendő atomok
|
| MASTER | Különféle rekordok száma, ellenőrző összegek
|
| END | Vég
|
- Sok probléma: bonyolult formátum, számos hibalehetőség, programmal
nehezen beolvasható (sokféle ellenőrzés kell, stb.), kémiai gráfot nem
mindig képes pontosan megadni
- Előny: ember által is jól olvasható
- Hidrogénatomok csak NMR-szerkezeteknél, ezekben több szerkezet is lehet
- mmCIF (macromolecular Chemical Interchange Format)
- bonyolult formátum, sokféle kulcsszóval, számos plusz információval
(pl. kötésszögek)
- ember számára nehezen olvasható, programokkal könnyen
- PDB formátum számos hibáját kiküszöböli
- MMDB
- ASN.1 szerkezet: hierarchikus felépítésű fájl. Ember számára nehezen,
számítógép számára könnyen olvasható
- Oldallánc-könyvtár alapján pontosan definiálja a kémiai gráfot
- NCBI használja, az ő Cn3D nézegetőprogramjuk ismeri
Összefoglalások a PDB-szerkezetekről, rengeteg linkkel más adatbázisokhoz
- A homooligomereknél a PDB általában csak egy alegységet tartalmaz
- PQS: automatikus módszerekkel generálják a biológiailag aktív oligomer
szerkezetet (nemtriviális, nem mindig egyértelmű, a valószínű szerkezetet
adja meg)
Nézegetők
Rasmol, Cn3d, Weblab Viewer, stb. (ingyenes programok): megjelenítik a
térszerkezeteket
Tökéletlenségek: pl. Rasmol az alternatív atompozícióknál hibázik (ld.
5hvp, C lánc)
Fehérjecsaládok adatbázisai
Klaszterezés
- Klaszterezés: eljárás, mellyel egy halmaz elemeit egymáshoz való közelségük alapján
csoportokba soroljuk. Lehet egyszerű vagy hierarchikus:
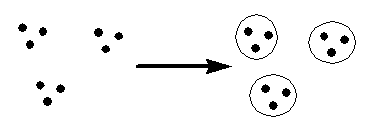
Egyszerű klaszterezés: részhalmazokra bontás
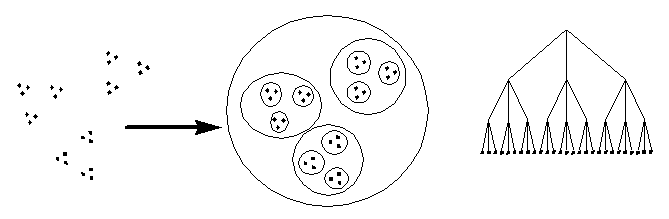
Hierarchikus klaszterezés: az osztályozás fastrukúrával
reprezentálható
- Bármilyen halmazon végezhető, amelyben két elem közötti távolság
értelmezhető.
- Szekvenciákra alkalmazva: szekvenciacsaládok adatbázisai
- Térszerkezetekre alkalmazva: szerkezeti családok adatbázisai
Szekvenciacsaládok adatbázisai
- Klaszterezésével családokba sorolhatóak a szekvenciák
- Sok ilyen adatbázis (CLUSTR, PROT-FAM, stb.)
- Bővebben egy másik előadásban
Szerkezeti családok adatbázisai
A fehérjék (klaszterezéssel) szerkezeti családokba sorolhatóak, a PDB
15000 szerkezete között csak kb. 600 teljesen különböző alapszerkezet (fold)
van.
A fold ("gomboly") fogalma
- Fold: Egy fehérje nagybani, durva szerkezete, a polipeptidlánc
gerincének durván vett térbeli lefutása. Magában foglalja a másodlagos
szerkezeti elemek körülbelüli, relatív elhelyezkedését és
összeköttetéseik sorrendjét. A folding (felgombolyodás)
szóból. Magyarul gombolynak mondhatnánk.
- A hasonló szerkezetű fehérjéknek ugyanaz a foldja, vagyis a gombolya. A
fold tehát egy fehérjecsaládot határoz meg, szerkezeti hasonlóság alapján.
Példa:
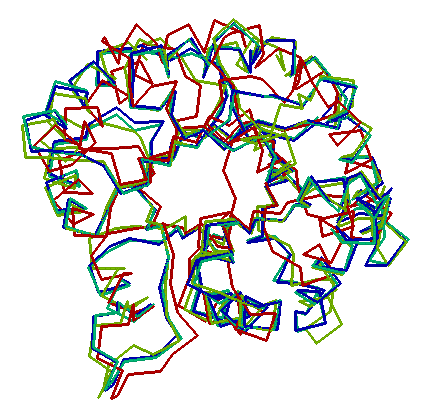
| 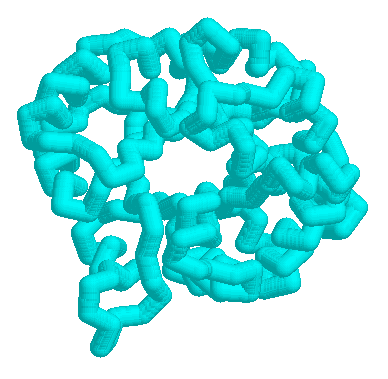
|
| Különböző fajokból származó trióz-foszfát izomerázok
(TIM) és hasonló fehérjék egymásra illesztett szerkezete (alfa-szénatomokból
álló váz).
| A fehérjecsalád fold-ja, azaz "gombolya", az ún.
TIM-barrel fold
|
- Részben manuálisan, részben automatizált módszerekkel készített adatbázis
- Hierarchikus rendszerezés:
- Fold ("gomboly"): Jelentős szerkezeti hasonlóság az
egyazon foldhoz tartozó szerkezetek között.
Lényegében ugyanolyan másodlagos szerkezeti elemek, ugyanolyan sorrendben,
ugyanolyan topológiával. A perifériális részek jelentősen eltérhetnek. Nem
feltétlenül közös eredet magyarázza a hasonló szerkezetet.
- Főcsalád (superfamily): Valószínűsíthető közös evolúciós
eredet az egyazon főcsaládhoz tartozó fehérjék között
Alacsony szekvenciaazonosság, de a funkcionális és szerkezeti hasonlóságok
közös evolúciós eredetre utalnak
- Család: egyértelmű evolúciós rokonság az egyazon családhoz
tartozó fehérjék között
A szekvenciaazonosság a család tagjai között 30% vagy nagyobb, vagy pedig a
hasonló funkcióból és szerkezetből egyértelmű az evolúciós rokonság
(alacsonyabb szekvenciaazonosság esetén is)
CATH adatbázis
- A hierarchia szintjei: Class, Architecture, Topology, Homologous
superfamily: Osztály, Architektúra, Topológia, Homológ főcsalád
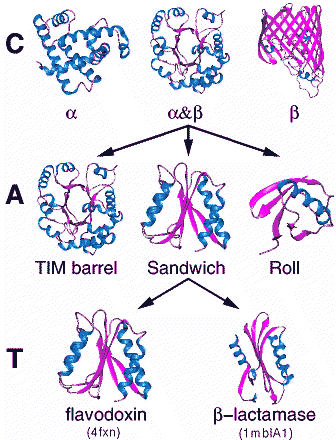
- Szintén részben manuálisan, részben automatikusan létrehozott adatbázis
- CATH hierarchia teteje: osztályok (class):
- Tisztán alfa fehérjék
- Tisztán béta
- Alfa és béta (a/b) (parallel béta, váltakozó alfa és béta régiók)
- Alfa és béta (a+b) (antiparallel béta, elkülönülő alfa és béta
régiók)
- Többdoménes fehérjék (domének más-más osztályban)
- Membrán- és sejtfelszíni fehérjék és peptidek
- Kis fehérjék (nagy része ligandum vagy prosztetikus csoport)
- "Coiled coil" fehérjék
- Kisfelbontású fehérjék
- Peptidek
- Tervezett fehérjék
- "CATH-kerék": az adatbázisban lévő osztályok megoszlása:
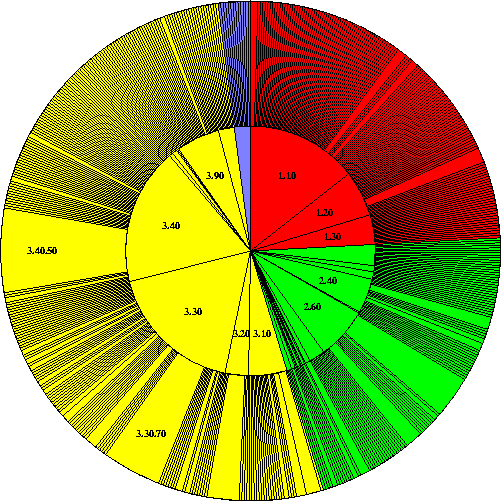
- Színek: piros (alfa), zöld (béta), sárga (alfa/béta), kék (nincs
másodlagos szerk.)
- Belső kerék: architektúrák
- Külső kerék: topológiák
Fontosabb architektúrák:
Tisztán alfa osztály:
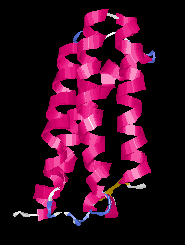
| 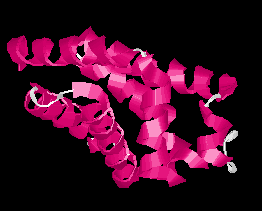
|
| Hélixköteg (citokróm C)
| Hélixek (hemoglobin)
|
Tisztán béta osztály:
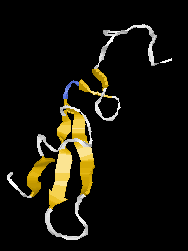
| 
| 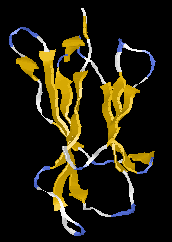
|
| Egyszeres redő (heregulin alfa)
| Béta hordó (porin)
| Béta szendvics (hisztokomp. antigén)
|

| 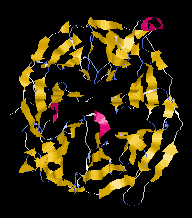
| 
|
| Béta hasáb (agglutinin)
| Béta propeller (metilamin dehidrogenáz)
| Béta szolenoid (Fágfeherje)
|
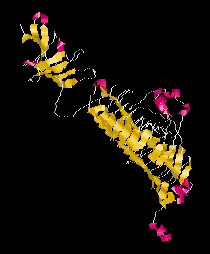
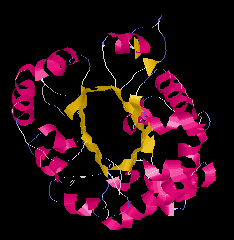
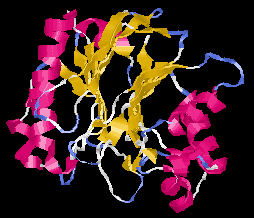
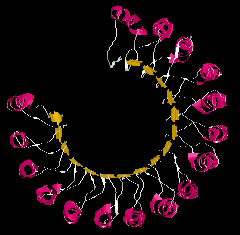
Alfa/béta és alfa+béta fehérjék:
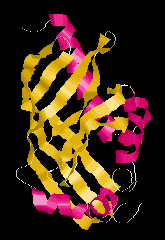
| 
| 
|
| Alfa-Béta tekercs (scytalone dehidratáz)
| Alfa-Béta hordó (trióz-foszfát izomeráz)
| Alfa-Beta 2-szendvics (barnáz)
|
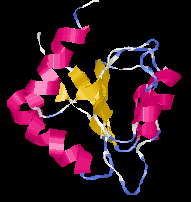
| 
| 
|
| Alfa-Béta 3-szendvics (génszabályozó fehérje)
| Alfa-Béta 4-szendvics (DNáz)
| Alfa-Béta Lópatkó (RNáz inhibitor)
|
- Teljesen automatikusan létrehozott adatbázis
- A DALI algoritmus a PDB-ben lévő szerkezetek között hasonlóságot állapít meg, ennek
alapján családokat különít el
- Előnye a SCOPpal és CATHtal szemben: a DALI szervernek el lehet küldeni
új, ismeretlen szerkezetet, s megtalálja a hozzá hasonlókat
A szerkezeteknek a SCOP, a CATH és az FSSP szerinti osztályozása
lényegében megegyezik, apróbb eltérésekkel. A legtöbb kiegészítő információ
a SCOP-ban található.